微生物是環境中的關鍵角色之一,也是生態系統中重要的一環,
了解微生物群落多樣性是目前大家十分感興趣的課題。
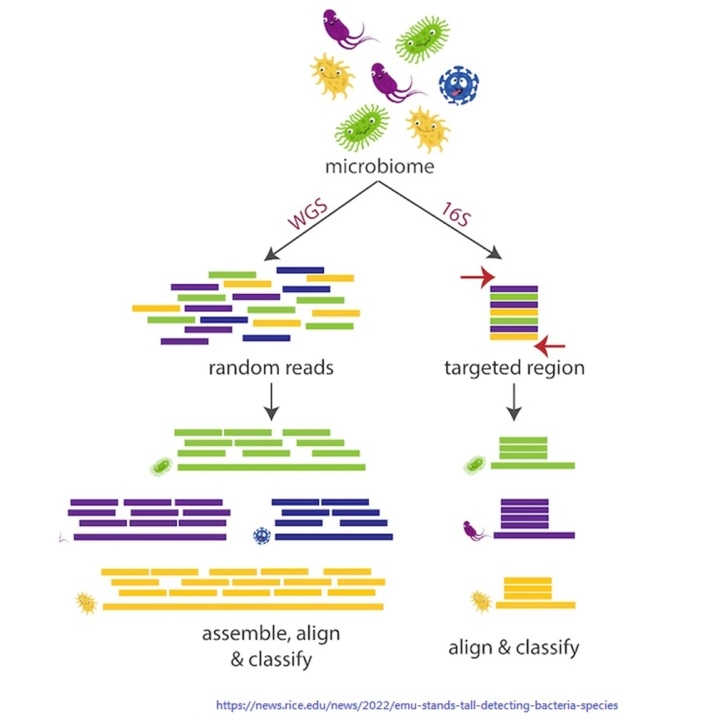
之前介紹過總體基因體學(metagenomics)是指環境中所有生物遺傳物質的總和,
今天要來介紹另一個metagenomics的NGS應用-- whole genome metagenomics
之前提過的16S metagenomics是只針對特定的區域(16S rDNA)進行定序,
只提供細菌微生物群落分類組成的資訊,適用於想了解物種的分布及多樣性的研究上使用
Whole genome metagenomics則是針對樣本中所有的微生物遺傳物質(DNA)進行定序,
適用於深入探討環境微生物之種類分布、生化代謝功能分析、物種與功能的協同作用(synergy),
方法是將樣本中的所有DNA萃取出來後,
以酵素剪切或物理性碎裂成小片段後進行建庫(Illumina/Ion Torrent)上機;
或是直接建庫後使用Oxford Nanopore或PacBio平台進行上機,
下面為大家整理出whole genome metagenomics的優缺點,
可以更清楚了解whole genome metagenomics的方法特性喔!
優點:
- 具有較高的解析度,可以辨識到種或菌株的物種層級
- 可提供基因功能信息
- 質體(plasmid) DNA也能定序到
- 細菌以外的物種也能辨識到,如:病毒、古細菌、真菌、藻類......
- 沒有PCR產生偏差(bias)的問題
缺點:
- 定序成本較高
- 容易受宿主DNA干擾
- 定序結果分析的困難度較高
#詳盡的售後服務諮詢
#專屬的數據管理系統
#迅速的發送結果報告
#專業的生物資訊分析
#源資生技
#TriIBiotech
#源資定序
#次世代定序
#Wholegenomemetagenomics
#IlluminaMiseq
# Nanopore
#Metagenomics
NGS & 第三代定序服務
Nanopore,Illumina長短序列機型齊全
高效率,高品質的好選擇
專業的樣品品管程序與文庫製備
專業的生物資訊分析與服務諮詢