在進行NGS定序之前,樣本需要先製備成Library文庫,而Library QC是「Library」建立後所進行品質檢測 (Quality Control, QC)的方法。目的是為了確認Library文庫的品質與完整性、文庫片段大小是否在預期範圍、文庫有無降解或污染以及文庫正確濃度 (過濃或過稀都會影響定序時cluster與data生成)。
以下為常見的NGS Library QC方法:
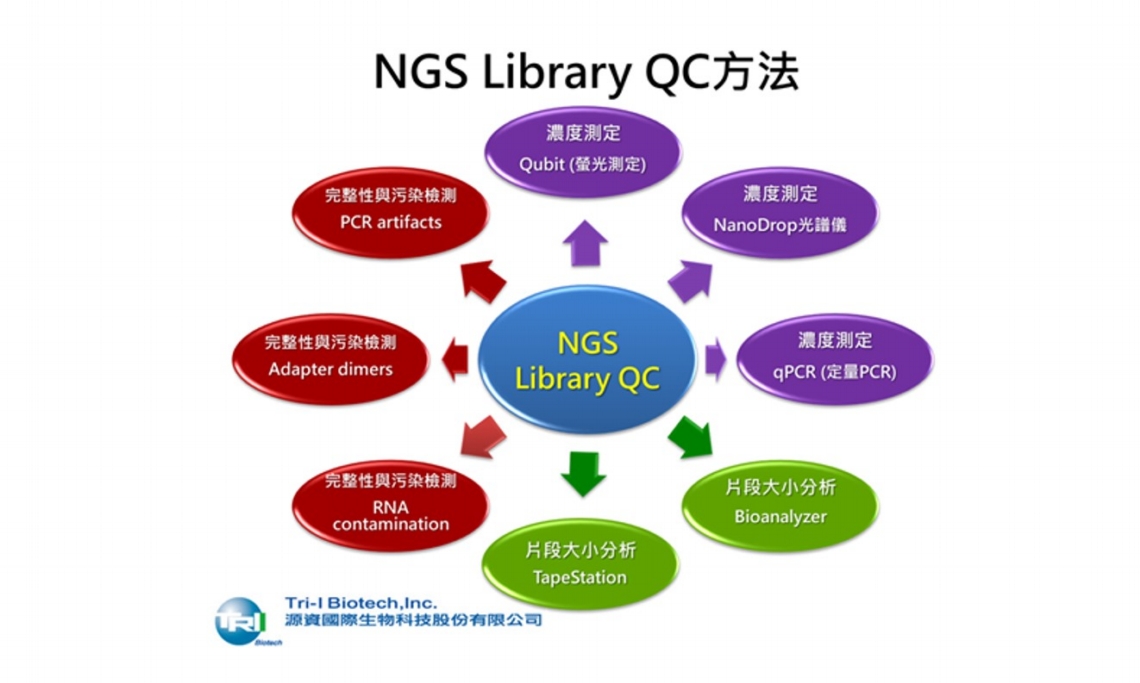
1. 濃度測定 (Quantification):
(1) 螢光測定 (Qubit)
優點:精確測量雙股DNA濃度,低濃度也準確。
缺點:不提供片段大小資訊。
(2) 分光光度計測定 (NanoDrop)
優點:快速、簡便。
缺點:容易受RNA或其他污染物干擾,不夠精確。
(3) 定量PCR (qPCR)
優點:可測序列ligation後的有效library濃度。
缺點:成本較高,需標準曲線與專用試劑盒。
2. 片段大小分析 (Size Distribution):
(1) Bioanalyzer (Agilent)
儀器:微流體晶片分析,常見於小量樣本。
特點:精確度高、圖形輸出直觀,費用中等。
(2) TapeStation (Agilent)
儀器:類似Bioanalyzer,但 throughput較高。
特點:自動化程度高,耗材成本較高。
* 片段大小分析重點:圖形呈現通常希望單一主峰、有預期片段大小 (例如300–500 bp)、無明顯primer dimer或過多小分子片段。
3. 完整性與污染檢測:
(1) RNA 殘留:使用Bioanalyzer的RNA分析或NanoDrop A260/A280比值觀察。
(2) Library adapter dimers:大小約120–150 bp,應該被最小化或移除。
(3) 非專一性PCR產物 (PCR artifacts) :檢測是否有非特異性擴增。
總結來說:
1. 進行NGS library QC是確保高品質數據與降低失敗率的重要步驟。建議多種方法搭配使用,例如:定量 (qPCR)+片段分析 (Bioanalyzer)進行雙重確認。
2. 一般常見QC合格標準建議:
(1) Library濃度 (qPCR):>10 nM (依定序平台可能不同)。
(2) 平均片段大小:約300–500 bp (Illumina平台常見)(依實驗目的可能不同)。
(3) 無明顯雙峰或dimer:單一主峰為佳 (依實驗設計可能不同)。
(4) 濃度偏差CV值 (多樣本間):<20%為理想。
# NGS_Library_QC
# Qubit
# NanoDrop
# qPCR
NGS & 第三代定序服務
Nanopore,Illumina長短序列機型齊全
高效率,高品質的好選擇
專業的樣品品管程序與文庫製備
專業的生物資訊分析與服務諮詢