單細胞RNA定序(Single cell RNA Sequencing; scRNA-seq)是一種利用次世代定序的方法對單一細胞mRNA進行擴增及定序的技術。
單細胞RNA定序的原理在此介紹的是使用微流體裝置進行油滴乳化封裝(oil emulsion encapsulation)方法產生微滴(droplet):
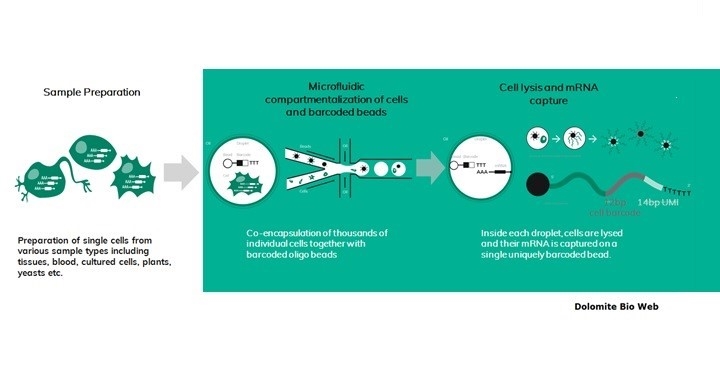
如下圖所示,裝置有三個通道:分別為細胞(Cell)、微粒(microparticle with lysis Buffer)、油(Oil),之後利用微流體的技術讓細胞與微粒先匯合,
再流經含有油的通道時,細胞微粒混合體就會被油滴一個一個的包埋,
以一個細胞搭配一個微粒的組合形成各自獨立的微滴(droplet)。
含有Poly A的mRNA細胞先與裂解緩衝液(Lysis buffer)中鑲嵌著DNA primer的微粒(microparticle)會合,
其結構為包含poly T、Cell barcode、UMI barcode以及PCR handle fragment之微粒
此時將細胞裂解,其中所含mRNA會釋出,此droplet內的微粒會與mRNA結合(PolyT 與mRNA的 polyA)
隨後打破微滴,再進行反轉錄RT、cDNA PCR、及Library文庫的程序。
文庫完成之後,每個細胞的library都會包含不同的barcode序列,
定序分析時便可透過這些序列去區分單一細胞或多細胞、
以及計算單一細胞中的RNA表現狀況。
欲知關於Single Cell數據分析的內容,
請見下回分曉~~
#Single-Cell-Sequencing
#Nadia-Instrument
#RNA-Sequencing
#源資國際生物科技股份有限公司
#TriIBiotech
NGS & 第三代定序服務
Nanopore,Illumina長短序列機型齊全
高效率,高品質的好選擇
專業的樣品品管程序與文庫製備
專業的生物資訊分析與服務諮詢