近年來有關單細胞轉錄體定序(scRNA-Seq)的研究越來越盛行,此技術能夠針對不同細胞群分析其基因的表現量差異。分析scRNA-Seq資料的方法,主要以二代定序的雙端數據資料為主。
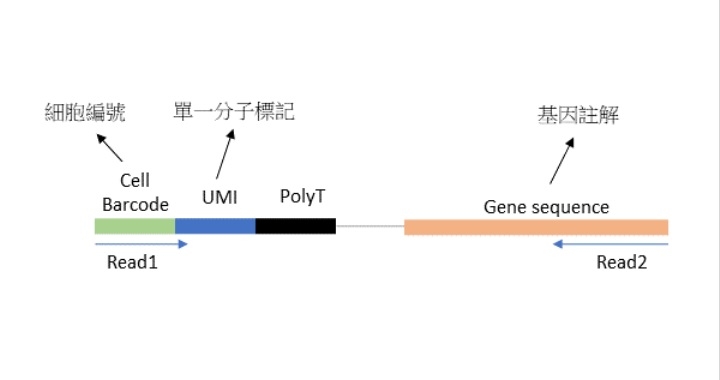
所獲得的定序資料結構如下圖所示。由於是雙端定序數據,通常其中一端序列資訊主要目的為辨識區分每單一細胞內的RNA分子,分成兩個部分:每顆細胞的辨識編碼(Cell Barcode)與RNA單一分子編碼(UMI, Unique Molecular Index);而另一端序列主要為RNA的基因序列,用來判斷是哪個基因。最後結合這兩端的資訊來獲得不同顆細胞之間內的基因表現量。
分析時會先去除定序品質較差的部分,以確保後續分析的準確性。之後,分成兩部分,一為包含細胞資料的那端序列,會被切割成Barcode及UMI兩個區塊進行分析。Cell Barcode的作用為區分每一顆細胞;而UMI的作用主要可以用來校正因PCR步驟導致的重複序列。而另一端RNA序列則會針對參考序列(Reference)進行比對,對序列進行基因註解。透過現有的軟體,如Cell Ranger或是dropSeqPipe就能完成上述的分析步驟。之後可以針對獲得的基因表現量數據結果,推薦使用Seurat軟體進行分析,針對基因表現量或特定基因的marker進行細胞分群,進而找出不同細胞群之間的差異基因等。
#詳盡的售後服務諮詢
#專屬的數據管理系統
#迅速的發送結果報告
#專業的生物資訊分析
#源資生技
#TriIBiotech
#scRNASeq
#SingleCellSequencing
NGS & 第三代定序服務
Nanopore,Illumina長短序列機型齊全
高效率,高品質的好選擇
專業的樣品品管程序與文庫製備
專業的生物資訊分析與服務諮詢