之前我們介紹過關於DNA甲基化,今天則來談談發生在RNA中的甲基化
RNA的甲基化會發生在腺嘌呤(adenine)的N-6 (m6A)、腺嘌呤的N-1 (m1A)及胞嘧啶(cytosine)的C-5 (m5C),其中最常見的就是m6A了
mRNA的5' Cap UTR甲基化作用為維持mRNA穩定、pre-mRNA加工 (processing)、mRNA的運送及轉譯 (translation)的起始等;
3' polyA的甲基化則有助於mRNA輸送出細胞核、轉譯的起始及polyA binding protein共同維持mRNA結構穩定。
除了mRNA之外,non-coding RNA (如lncRNA、miRNA、tRNA、rRNA..等)中也存在許多甲基化修飾。
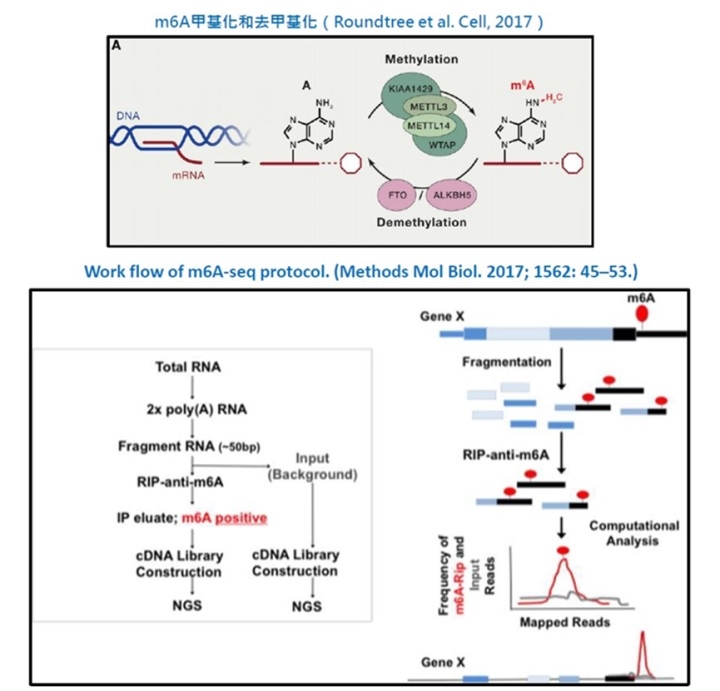
RNA甲基化的過程會有三類酵素參與其中:
1. Writers: 將甲基化修飾寫入RNA鹼基中,如METTL3、METTL14與WTAP會形成複合體來催化RNA的m6A甲基化
2. Erasers: 將RNA的甲基化信號移除,如FTO和ALKBH5
3. Readers: 辨識及讀取RNA甲基化的訊息,並參與下游RNA的轉譯及降解等過程,如YTHDF家族的蛋白等
RNA甲基化要如何偵測呢?
目前所使用的方法為針對m6A所發展出的MeRIP-Seq (m6A RNA immunoprecipitation,或m6A-Seq),
先將帶有polyA的mRNA進行碎裂後,其中一半的樣本做為控制組繼續一般文庫建構;
另一半的樣本則使用m6A抗體的磁珠進行富集並回收含有m6A的mRNA片段,
接著完成m6A-Seq建構文庫後,與控制組的RNA-Seq文庫分別進行二代測序上機。
這個方式得到的數據分析並不能達到單鹼基的分辨率,
只能夠鑑定mRNA上m6A甲基化程度較高的區域,解析度約為100 bp左右。
三代測序的發展,在甲基化檢測上的應用可以克服二代測序的阻礙,
如同之前在DNA甲基化中所提及的利用PacBio或Oxford Nanopore平台,
樣本不需任何前處理作用,直接利用甲基化與未甲基化鹼基的特性不同進行定序。
並且可以針對沒有Reference序列的樣本進行甲基化位點分析;
雖然以往三代定序的缺點為錯誤率較高,
但是最新發展的三代定序平台已有大幅提升正確率的趨勢,
看來三代定序平台的應用在日後勢必會愈來愈廣。
#詳盡的售後服務諮詢
#專屬的數據管理系統
#迅速的發送結果報告
#專業的生物資訊分析
#源資生技
#TriIBiotech
#NGS
#甲基化methylation
#m6A
NGS & 第三代定序服務
Nanopore,Illumina長短序列機型齊全
高效率,高品質的好選擇
專業的樣品品管程序與文庫製備
專業的生物資訊分析與服務諮詢