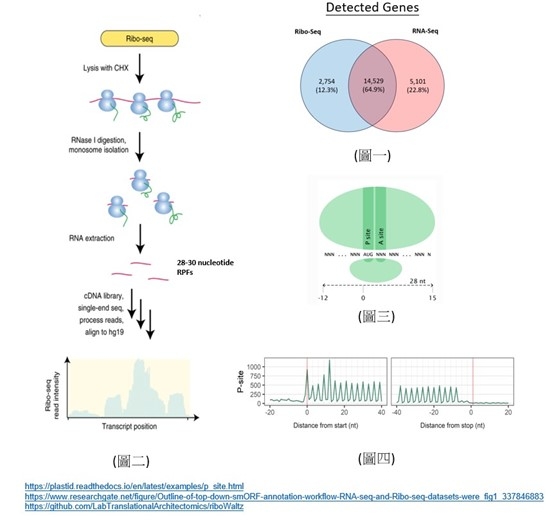
Ribo-Seq近幾年來開發的一種技術,常用於評估mRNA轉譯的蛋白質合成速度及控制機制。雖然全基因表達的分析中,通常使用RNA-Seq的方法來確認RNA的表現結果來取代分析蛋白質合成,但最終蛋白質表現的多寡並不總是與mRNA表現量一致 (圖一),而這是由於mRNA還會進行調控及修飾等調節機制。而Ribo-Seq就成為評估蛋白質合成速度和研究轉譯與控制機制的主要方法。Ribo-Seq是基於對核醣體mRNA結合位點(Ribosome protected mRNA footprinting, RPF)進行RNA-Seq分析 (圖二)。由於每個核糖體結合位點代表一個核糖體的位置,它們不僅顯示了哪些 mRNA表現,而且還揭示了這些 mRNA 的 CDS 被轉譯的部位,這能夠用於繪製體內正在作用中轉譯核糖體的圖譜,以提供有關轉譯暫停、停滯和轉譯起始位點等訊息,以及蛋白質數量的估計。
由於序列片段普遍偏小,通常只拿單端的序列進行分析。定序所獲的數據先移除adapter、RPF及quality低的序列,以確保後續分析的準確度。之後針對rRNA及tRNA進行Mapping並保留沒有比對到的reads。之後再針對參考序列進行Mapping的分析,後續如RNA-Seq的分析相似,可以進行基因之間表現量的差異比較,Heatmap的繪製、NMDS及PCA的分群繪製、Gene Ontology及Pathway的分析等。此外,透過位於參考序列上的Mapping情況,Ribo-Seq的結果可以額外偵測到蛋白質轉譯的可能起始位置 (P-site) (圖三、四)。
NGS & 第三代定序服務
Nanopore,Illumina長短序列機型齊全
高效率,高品質的好選擇
專業的樣品品管程序與文庫製備
專業的生物資訊分析與服務諮詢