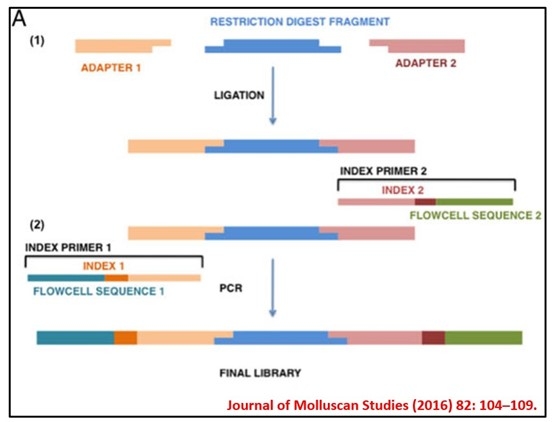
ddRAD-seq (double digest Restriction-site Associated DNA sequencing),是一種簡化基因組定序技術,基於選用兩種限制酶來針對基因組作裁切,只定序裁切後符合預期長度的片段,以達到降低全基因組定序成本之目的;並藉由減少定序的資訊量,來提高有效 SNP (Single Nucleotide Polymorphism)位點的篩選效率,有利於基因分型和遺傳變異數據的統整。
相較於單一限制酶作用輔以超音波碎裂的傳統 RAD (300-700bp),ddRAD所得DNA 片段大小會比較集中 (~500bp),有助於建庫時解序片段的篩選。此外,ddRAD搭配雙酶切所使用的雙端 adapter 可使用UDI(Unique Dual Index),相對於只有單端 index 的 RAD,除了避免了 index hoping 的問題,於大樣本量的群體研究,可有效地減少掛載不同 index adapter 的使用及合成成本。
而源資實驗室採用可銜接適配Illumina文庫製備套組的ezRAD-seq,此方法學承接了ddRAD雙酶切的優點,且精簡統一的實驗步驟降低了文庫建構前置作業的複雜性,除了可減少試驗偏差,也大大地加快了提供分析數據的時間。
小編幫大家整理ddRAD-seq的優點:
- 成本效益:只定序裁切後符合預期長度的部分基因組,有效地降低定序成本。
- 降低基因組複雜性:透過限制酶裁切特定位點的特性,降低了基因組整體的複雜性,有利於大型或複雜基因組生物的定序和分析。
- 適用性廣:可應用於各種無論有無參考基因組 (reference genome)的生物體,對於非模式物種的研究幫助非常大。
- 可規模化:此方法學可高度規模化,可同時應用於大量個體,使其適合需要大樣本量的群體基因組學研究。
- 減少偏差:由於分析方法學不依賴先前的序列資訊,因此減少了使用參考基因組引入的偏差 ,使其更適合非模式物種的研究。
- 再現性高:使用特定的限制酶可確保不同處理樣本會針對相同的基因組區域進行定序,從而提高分析結果的再現性。
NGS & 第三代定序服務
Nanopore,Illumina長短序列機型齊全
高效率,高品質的好選擇
專業的樣品品管程序與文庫製備
專業的生物資訊分析與服務諮詢